〜ハイ・スループット微生物学のための自動コロニー表現型解析技術の進展〜
概要
寒天プレート上の固体培養は、単一クローンの分離と微生物集団のコロニー形成単位(CFUs)による定量化を可能にする、微生物学の基礎技術です。しかし、従来の技術は、手作業とエンドポイント測定に依存しているため、スループットが低く、ラグタイムや増殖率のような重要な増殖動態データが欠如していました。特に、数百万の遺伝子変異体をスクリーニングする必要がある合成生物学や微生物工学において、これは重大なボトルネックとなっています。
本ホワイトペーパーでは、自動化された固体培養増殖モニタリングシステムの進歩に焦点を当てます。これらのシステムは、タイムラプス技術(ScanLag、ColTapp他)から、可視コロニーが形成される前にマイクロコロニーの光学密度 (OD)を検出する次世代の Bacterial Growth Monitor (BGM) へと進化しています。これらの技術革新は、伝統的なCFUアッセイを定量的でハイ・スループットなツールへと変貌させ、稀で高性能な変異体の客観的な識別と回収を可能にします。
1. 固体培養の価値と従来の限界
1.1 固体培養の決定的な利点
液体培養は培養液中に存在する様々な菌株集団の平均のパラメーター(濃度、増殖速度等)を提供しますが、固体培養は空間的分離という決定的な利点を提供します。これは、個々のクローン集団の独自の表現型特性を観察するための前提条件です。
1.2 伝統的なCFUアッセイの定量的限界
従来のCFUアッセイは、プレートへの播種、インキュベーション、手動での計数によるエンドポイント測定に限定されます。
- 時系列データの欠如: 最終的な総生存細胞数を決定するのみで、細胞が時間とともにどのように増殖し、コロニーを形成したかについての時系列的洞察がありません。このために、菌株のロバスト性を示すラグタイムや増殖率の測定ができません。
- 主観的な表現型解析: コロニーサイズ、テクスチャ、色などの特性は、一般的に定性的に記録され、高性能な変異体と野生型との正確な比較ができません。
- 高密度での計数エラー: 大規模スクリーニングのための高密度播種ではコロニーが頻繁に融合し、手動および古い自動化システムでは、これらの融合領域が単一として誤ってカウントされ、真のCFUカウントが著しく過小評価されます。
これらの制約により、従来の固体培養法は、迅速な遺伝子最適化に継続的かつ定量的なフィードバックが必要なDBTL (Design-Build-Test-Learn)サイクルに用いるには不十分です。
2. 自動化された固体培養増殖モニタリングの進展
最新の自動化システムは、インキュベーター内蔵のスキャナー/カメラと高度なコンピュータービジョンを統合し、非侵襲的で高解像度のモニタリングを実現します。これにより、数千もの個々のクローンの増殖動態を同時に追跡できます。
2.1 タイムラプスシステム
初期の自動化モニタリングは、手動のエンドポイントカウントをコロニー増殖のタイムラプス時系列解析に置き換えました。
- ScanLag: 非侵襲的な周期的画像取得を通じてコロニー増殖曲線を追跡し、適応時間(ラグタイム)とコロニー増殖率の二次元分布を特徴づける実用的な方法を提供しました。
- ColTapp: 最新の画像処理によりコロニー増殖とラグタイムのダイナミクスを正確に定量化し、抗生物質耐性(persistence)の研究などに利用されています。
2.2 次世代モニタリング:プレ・コロニー検出
最も進んだシステム、例えばCarbGeMが開発したBacterial Growth Monitor (BGM)は、反射光ではなく透過光を検出するための高解像度センサーアレイを使用しています。
- マイクロコロニーOD測定: BGMは、レンズなしの1,000 x 1,000ピクセルの高空間分解能と5分ごとの高時間頻度で、固体培地上の光学密度 (OD)を測定します。
- 検出限界の向上: わずか数個の細胞のクラスター(マイクロコロニー)による光散乱の微妙な変化を測定することで、物理的なコロニーが形成される前に増殖の開始を検出できます。この能力は、アッセイ時間を数時間短縮します。
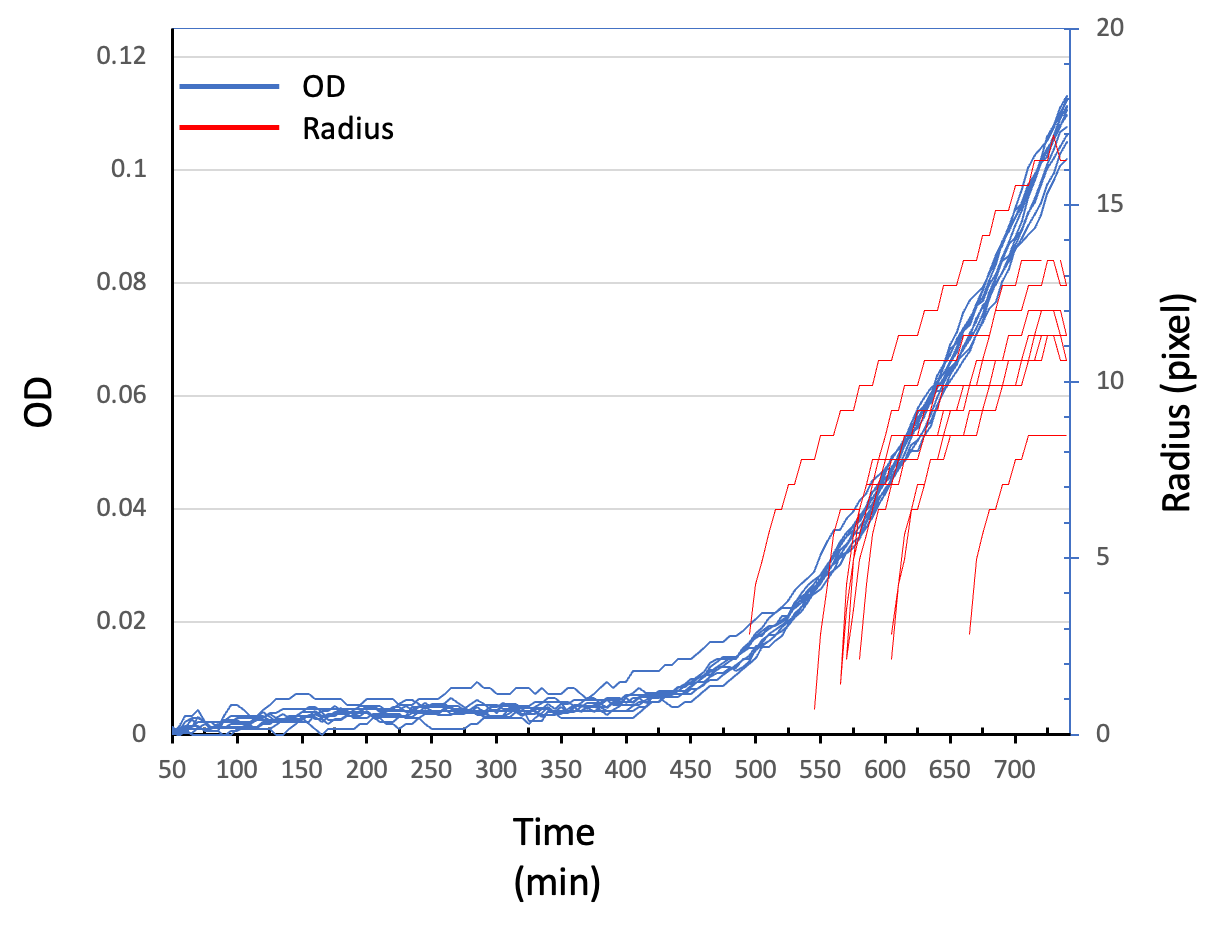
- 比較例: この比較実験では、BGMとカメラ装置は同じインキュベータ内に設置されました(Figure 1a)。BGMが400-450分でODの増加を検出したのに対し、カメラは500-550分でコロニーを検出しました(Figure 1b参照)。
Figure 1 (a) 同一インキュベーター内のBGM(左)とカメラ装置(右)。(b) BGMが検出した成長速度上位10ピクセルのOD曲線(青)と、カメラ画像から検出した最初の10コロニーの半径曲線(赤)。
- 強化されたフェノミクス:データストリーム(数千の個々のコロニーにおける高解像度の時系列データおよびODデータ)は、機械学習モデルのデータセットを大幅に充実させ、阻害作用、毒性、および最終的な株の性能に関する予測精度を向上させます[3]。
2.3 ハードウェア統合とデータ駆動型の選択
高度な時系列データは、ロボティクスと統合されます。
- 融合コロニーの分解: MCountのような高度な画像処理アルゴリズムは、重複コロニーを正確に分離し、高密度播種における計数過小評価を修正します。
- データ駆動型の選択: モニターが提供する定量的な指標(ラグタイム、増殖率、プレ・コロニーOD曲線)は、ロボットコロニーピッカーと統合されます。これにより、例えば「速いラグタイムだがゆっくりと増殖するクローン」といった、特定のマルチパラメータプロファイルに基づいてクローンを客観的に選択し、スケールアップのために最良の変異体を回収することが可能になります。
3. 応用分野と影響
定量的な固体培養モニタリングへの移行は、バイオエンジニアリング・パイプラインの根本的なボトルネックを克服し、幅広い研究分野に影響を与えます。
3.1 微生物工学:フィットネスと生産の分離
このシステムの最大の利点は、「稀な変異体のボトルネック」を解決することです。
- 競合からの保護: 代謝負荷により増殖が遅い改変された菌株は、液体培養では競合により失われやすいですが、固体培養では空間的分離により各細胞がコロニー内で保護され、ゆっくりと増殖する高生産性の変異体も検出されます。
- 分離された表現型: コロニーの増殖動態(フィットネス)と表現型特性(例:製品相関色、マイクロコロニーOD)を個別に測定することで、研究者は両特性の最適なバランスを選択できます。
3.2 臨床および発見研究への応用
詳細な時系列データは、臨床および発見研究において重要です。
- 抗生物質の発見: 阻止円や増殖曲線をより早い段階で検出する能力は、新しい抗生物質、ペプチド、およびバクテリオファージの探索を大幅に加速します。
- 細菌の持続性: 個々のクローンの適応時間を正確に測定することは、慢性感染症を理解するために不可欠な、薬剤耐性の「パーシスター」メカニズムの研究に使用されます。
4. 結論
自動化された固体培養モニタリングシステムは、従来のCFUアッセイを成功裏にデジタル化し、定量化しました。ScanLag、ColTappから高度なBacterial Growth Monitorに至るこれらのプラットフォームは、個々のクローンに関するハイ・スループットで時間分解された時系列データを提供することで、手動スクリーニングの主観性と限界を排除します。この技術は、合成生物学と産業微生物学におけるクリティカルなボトルネックを解消するために必要な解像度を提供し、バイオマニュファクチャリングの成功に不可欠な、稀にしか発現しない特異な変異体の確実な識別と回収を保証します。
5. 引用文献
- Levin-Reisman, I., et al. (2014). ScanLag: High-throughput Quantification of Colony Growth and Lag Time. Journal of Visualized Experiments, (89):51456. link
- Bar, J., et al. (2020). Efficient microbial colony growth dynamics quantification with ColTapp, an automated image analysis application. Scientific Reports, 10, 16084. link
- Taketani, M., et al. (2023). Image Sensor-Based Real Time Monitoring of Bacterial Growth on Agar Plates. IDWeek, Boston.
- Molecular Devices. (2025). Clone Screening Solutions, Automated Colony Picking. link
- Chen, S., et al. (2025). MCount: An automated colony counting tool for high-throughput microbiology. PLoS One, 20 (3), e0311242. link
- Abbate, E., et al. (2023). Optimizing the strain engineering process for industrial-scale production of bio-based molecules. Journal of Industrial Microbiology and Biotechnology, 50(1), kuad025. link
- Jiang, Y., et al. (2023). Strain and process engineering toward continuous industrial fermentation. Frontiers of Chemical Science and Engineering, 17(6), 632–641. link
- Jin, C., et al. (2022). High-throughput identification and quantification of single bacterial cells in the microbiota. Nature Communications, 13(1), 896. link
- CarbGeM Inc. (2025). Bacterial Growth Monitor (BGM). link